

| Processo seletivo Mestrado/Doutorado (Genética - UFMG: http://pggenetica.icb.ufmg.br): Há vagas para alunos de doutorado e mestrado para projetos envolvendo organização, evolução e função de DNAs repetitivos. Contato: Prof. Gustavo Kuhn (gcskuhn@ufmg.br) |
|
Grande
parte (>50%) do genoma de muitas espécies de
eucariotos, incluindo mamíferos, insetos e plantas, é
composta por DNAs repetitivos, que podem ocorrem
dispersos no genoma, como os Elementos Transponíveis,
ou arranjados em tandem. Os DNAs satélites (satDNAs)
são os principais representantes dos DNAs repetidos em
tandem. São formados por milhares de cópias de
sequencias de DNA, organizadas uma após a outra, que
formam cadeias extensas que frequentemente alcançam
centenas de milhares de pares de bases. Várias
famílias de satDNAs podem coexistir em um mesmo genoma
e juntas podem representar mais de 30% do total do DNA
genîmico. Durante muito tempo, satDNAs foram
considerados como “DNA entulho”. No entanto, nos
últimos 10 anos, vários estudos vêm mostrando exemplos
de satDNAs envolvidos em diversas funções celulares,
como na manutenção de centrômeros, formação da
heterocromatina e expressão gênica. Apesar destas
associações funcionais, satDNAs estão entre os
componentes do genoma de evolução mais rápida. Estas
mudanças nos satDNAs, tanto qualitativas como
quantitativas, impactam não somente a arquitetura e
evolução de genomas, mas também processos evolutivos
que dão origem a novas espécies.
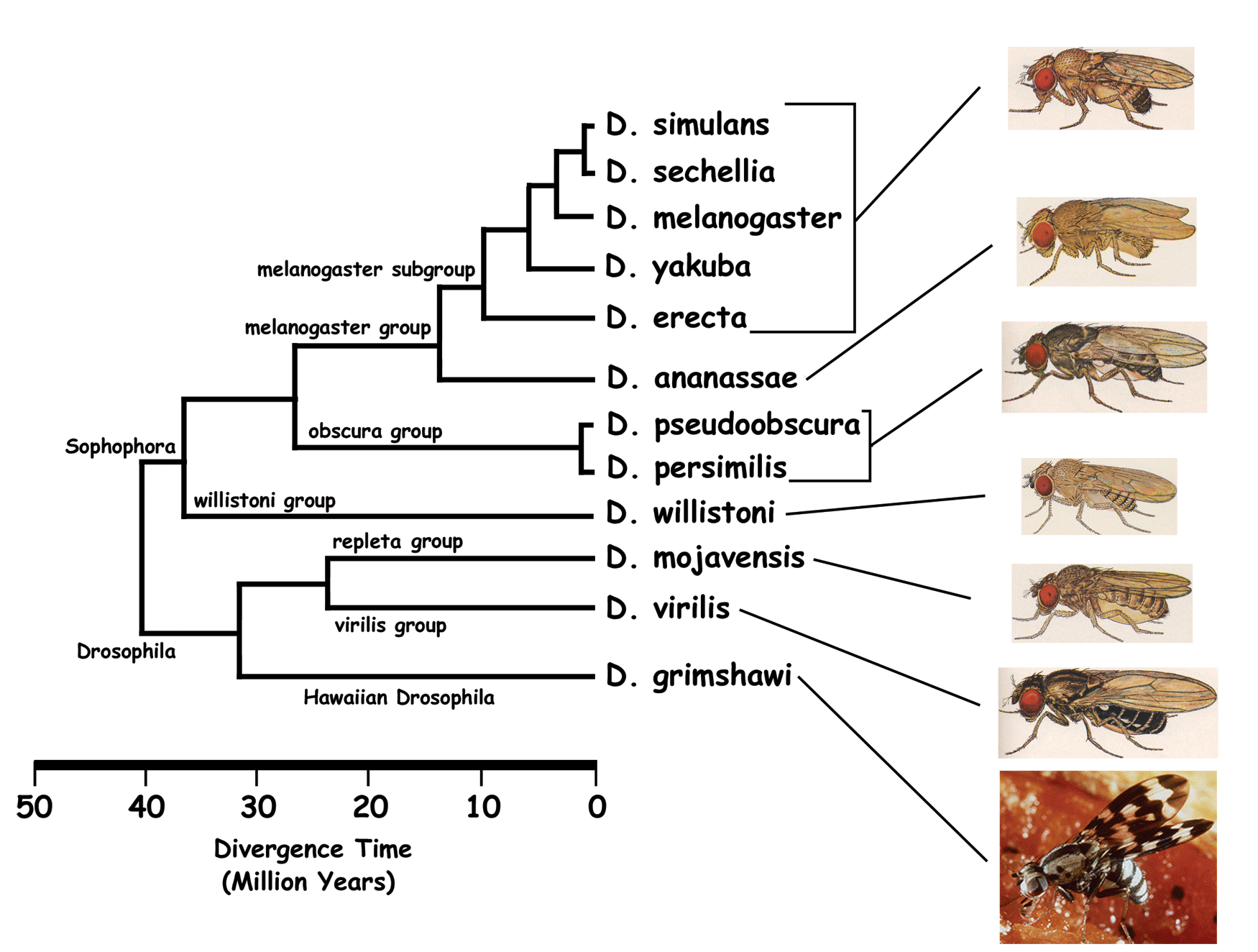
A introdução do sequenciamento de segunda geração (e.g. Illumina) possibilitou um avanço na identificação e estudo dos satDNAs de várias espécies de eucariotos. Mas por outro lado, a alta homogeneidade entre cópias de satDNAs, associada a longa extensão de suas cadeias, impedem a sua montagem através de reads curtas (~100 bp) de sequenciamento. Consequentemente, nosso conhecimento sobre os satDNAs, mesmo na era genômica, ainda é limitado em relação ao restante do genoma. Porém, nos últimos 5 anos, novas tecnologias de sequenciamento de terceira geração, como Pacbio ou Nanopore, conseguiram gerar reads longas de dezenas a centenas de kilobases. Com este recente avanço tecnológico, os primeiros trabalhos relatando a montagem completa de satDNAs estão sendo publicados e preenchendo uma lacuna científica histórica em estudos genômicos. Várias espécies de Drosophila tiveram seus genomas sequenciados recentemente através de uma combinação de sequenciamento Illumina (reads curtas) e Nanopore (reads longas). Projetos atuais de nosso grupo estudam os satDNAs destas espécies de Drosophila. Nossos estudos pretendem contribuir para um melhor entendimento de vários aspectos sobre os satDNAs, como sua origem, estrutura, mecanismo de amplificação, evolução e função.
Kuhn
GCS, Heringer P, Dias GB (2021). Structure,
organization, and evolution of satellite DNAs:
Insights from the Drosophila repleta and D.
virilis species groups. Prog Mol Subcell Biol.
60:27-56. doi: 10.1007/978-3-030-74889-0_2.
Nascimento AML, Silva BSML, Svartman M, Kuhn GCS (2020). Tracking a recent horizontal transfer event: The P-element reaches Brazilian populations of Drosophila simulans. Genet Mol Biol. 43(2):e20190342. doi: 10.1590/1678-4685-GMB-2019-0342. Silva BSML,
Heringer P, Dias GB, Svartman M, Kuhn GCS (2019).
De novo identification of satellite DNAs in the
sequenced genomes of Drosophila virilis and
D. americana using the RepeatExplorer and
TAREAN pipelines. PLoS One. 14(12):e0223466. doi:
10.1371/journal.pone.0223466. Dias BB, Heringer P,
Kuhn GCS (2016). Helitrons
in Drosophila: chromatin modulation and tandem
insertions. Mobile
Genetic Elements doi:
10.1080/2159256X.2016.1154638 Dias BB, Heringer
P, Svartman M, Kuhn GCS (2015). Helitrons shaping the
genomic architecture of Drosophila: enrichment of
DINE-TR1 in α and β-heterochromatin, satellite DNA
emergence and piRNA expression. Chromosome Res doi:10.1007/s10577-015-9480-x Guillén Y, Rius N, Delprat A, Williford A, Muyas F, Puig M, Casillas S, Ràmia M, Egea R, Negre B, Mir N, Camps J, Moncunill V, Ruiz-Ruano FJ, Cabrero J, de Lima LG, Dias GB, Ruiz JC, Kapusta A, Garcia-Mas J, Gut M, Gut IG, Torrents D, Camacho JPM, Kuhn GCS, Feschotte C, Clark AG, Betrán E, Barbadilla A, Ruiz A. (2015). Genomics of ecological adaptation in cactophilic Drosophila. Genome Biol Evol 7(1): 349-366. Dias GB, Svartman M, Delprat A, Ruiz A, Kuhn GCS (2014). Tetris is a foldback transposon that provided the building blocks for an emerging satellite DNA of Drosophila virilis. Genome Biol Evol 6(6):1302-1313. doi: 10.1093/gbe/evu108 Kuhn GCS, Kuettler H, Moreira-Filho O,
Heslop-Harrison JS (2012).
The 1.688 repetitive DNA of Drosophila: Concerted evolution at different
genomic scales and association with genes. Mol Biol Evol 29(1):7-11. doi:10.1093/molbev/msr173.
Link
por
PDF. |